⚠️ Cette page donne accès uniquement aux informations publiques d'une brique logicielle. Pour accéder aux autres contenus, merci de vous connecter ou d’adresser une demande à TRAIL afin d’obtenir un compte.
Sorbetto
Sorbetto in an open-source Library. Its development started at the TRAIL summer worshop 2025 in London (project team 3: Let’s Tile Together). It produced Tiles with a variety of flavors to analyze, compare and rank the performances of entities assimilable to two-class classifiers.
Learning for Optimizing
This repository contains the code used to research the application of reinforcement learning techniques for local search methods to solve combinatorial optimization problems.
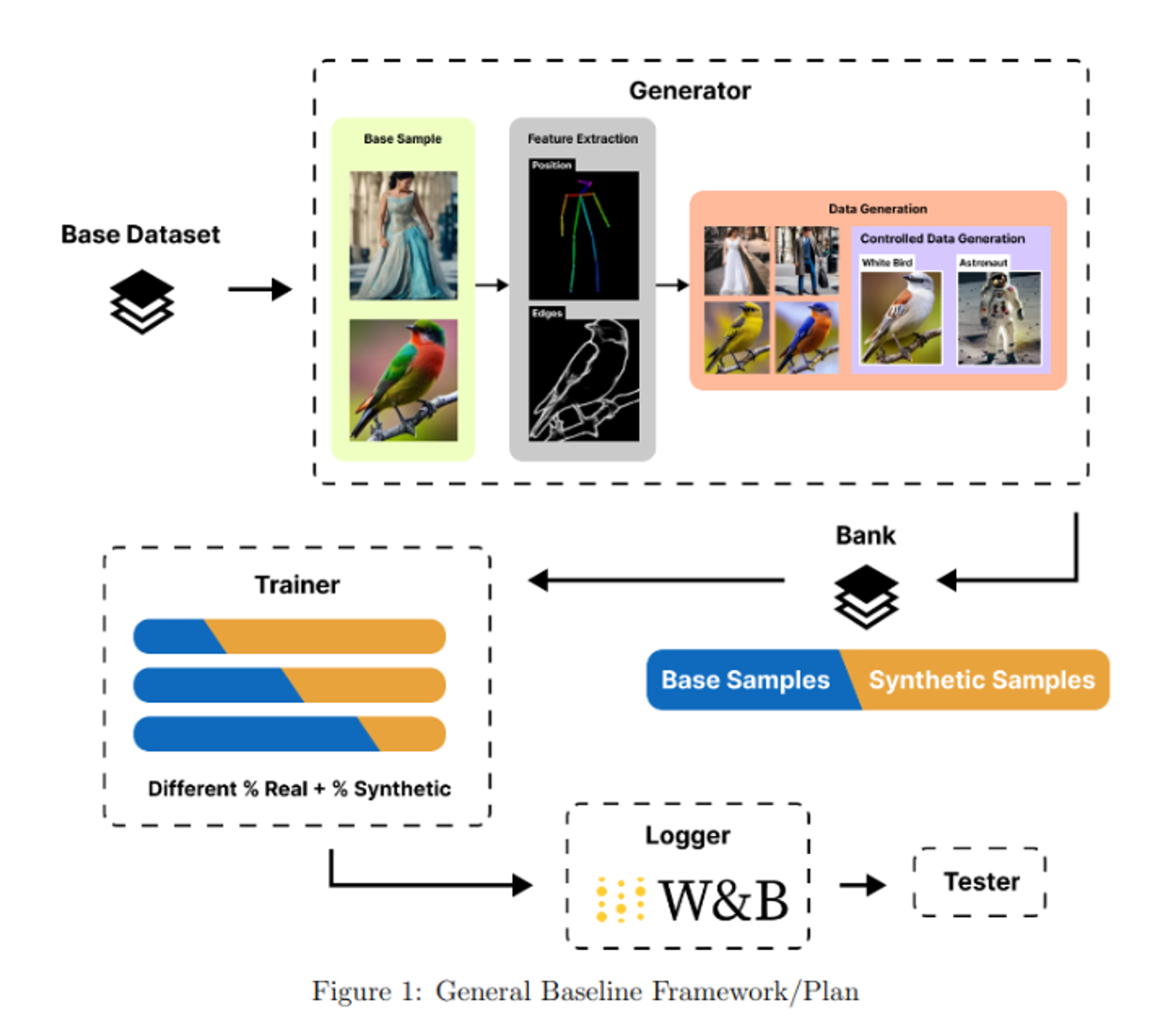
CIA : Data Augmentation Framework Using Stable Diffusion
Controllable Image Augmentation Framework based on Stable Diffusion :
This is a data generation framework that uses Stable Diffusion with ControlNet, to do Data Augmentation for Object Detection using YOLOv8. Models can be trained using a mix of real and generated data. They can also be logged and evaluated. This framework helps you train better models using data augmentation for data constrained scenarios.
Explainflix: Interactive Recommender System with LLM-Generated Explanations
Overview: Our project introduces an advanced interactive recommender system that combines graph-based collaborative filtering with Large Language Model (LLM)-generated explanations. Designed to enhance transparency and user trust, this system provides clear, context-rich explanations for recommendations, addressing a common issue with traditional methods.
Key Features:
- Graph-Based Collaborative Filtering: Employs graph-based techniques to refine and personalize recommendations based on user-item relationships and preferences.
- LLM-Generated Explanations: Utilizes state-of-the-art LLMs to generate detailed, engaging explanations for each recommendation, making the system's rationale more transparent and understandable.
- Real-Time Interaction: Offers a user-friendly web interface for quasi-real-time feedback and interaction, allowing users to engage directly with the recommender system and explore why an item was recommended.
Explainability Integration for Convolutional Neural Networks
This software brick is designed to introduce an explainability framework into convolutional neural networks (CNNs) used for binary classification, ordinal classification, or regression tasks.
By reshaping the network’s latent space, this tool allows for the exploration of any features of interest, providing insight into which features drive the model's final decisions.
MUSE - VSenseBox

VSenseBox is a small all-in-one Python toolbox filled with many vision/visual sensing modules such as object detectors and object trackers, etc. These modules are well integrated together and can be easily selected and configurated with minimal coding.
- Integrate with popular object detectors including YOLO v3, v4, v5, v8, v9, and more.
- Integrate with bbox trackers including Centroid, SORT, DeepSORT, and more TBA.
- Support Python 3.9-3.12 on Windows, Linux, and macOS.
- Support YAML config file for each individual module.
- Integrate with PyQt GUI for easy config.
This toolbox is a part of Win2Wal - MUSE project (Multimodal Sensing Environment for Mobile Applications), funded by Walloon Region, Belgium.
Segmentation of Histopathology Images Using SSL and AL with Missing Annotations
Real-world segmentation tasks in digital pathology require a great effort from human experts to accurately annotate a sufficiently high number of images. Hence, there is a huge interest in methods that can make use of non-annotated samples, to alleviate the burden on the annotators. In this work, we evaluate two classes of such methods, semi-supervised and active learning, and their combination on a version of the GlaS dataset for gland segmentation in colorectal cancer tissue with missing annotations. Our results show that semi-supervised learning benefits from the combination with active learning and outperforms fully supervised learning on a dataset with missing annotations. However, an active learning procedure alone with a simple selection strategy obtains results of comparable quality.
This work has been accepted at CVAMD 2023 (ICCV workshop), with the title "Computational Evaluation of the Combination of Semi-Supervised and Active Learning for Histopathology Image Segmentation with Missing Annotations". This work was done by Laura Galvez Jimenez, Lucile Dierckx, Maxime Amodei, Hamed Razavi Khosroshahi, Natarajan Chidambaran, Anh-Thu Phan Ho, and Alberto Franzin.
Temperature field prediction in additive manufacturing process
Additive Manufacturing (AM), also known as 3D printing, is a disruptive manufacturing technology that has grown rapidly in the manufacturing industry and has gained a lot of attention owing to its ability to manufacture parts with complex features by using a layer-by-layer approach. The variability in the final product quality is however one of the major hurdles to the widespread application of such techniques in production environment. In particular, the mechanical properties and the quality of the manufactured part largely depend on the distribution of temperature fields during the AM process. Numerical simulations, such as finite element analyses, are commonly used to simulate the thermal history during the AM process, but they are known to be expensive and time-consuming, and hence they cannot be used in real-time. In this context, we propose a machine learning approach for the fast and accurate prediction of the thermal field evolution. Our approach consists of a variational autoencoder that encodes the thermal fields into a latent space, combined with a recurrent neural network that simulates the temporal process in the latent space. In order to generate the datasets for training and testing this model, 256 finite element-based
thermal simulations were performed using Cenaero’s virtual manufacturing software, Morfeo.
ALAMBIC

ALAMBIC, ou Active Learning Automation with Methods to Battle Inefficient Curation, est une plateforme web open-source sous forme de docker pour l'étude et le développement de modèles d'apprentissage automatique ave l'aide de l'apprentissage actif.
Qu'est-ce que l'apprentissage actif ?
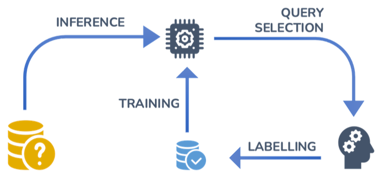
L'apprentissage actif est utilisé quand seule une portion des données totales est labellisée et que labelliser les données est coûteux en temps et/ou en prix. Cette méthode optimise la labellisation des données en sélectionnant les échantillons les "plus intéressants" pour être labelliser.
Le modèle, ici appelé l'apprenant, est initalement entraîné seulement avec la portion labellisée des données. Ensuite, il va essayer de prédire les labels de la portion non-labellisée des données. Avec ces prédictions, l'apprenant va sélectionner l'échantillon, aussi appelée la query, duquel il est le plus intéressé d'apprendre le vrai label. Ce processus est appelé la sélection de la query et peut-être faite en fonction de différentes strategies, comme par example en choisissant la query la plus représentative de la population des échantillons ou la query dont le modèle est le plus incertain de la prédiction.
La query est alors présentée à un oracle, la plupart du temps un humain, qui va la labelliser. Après ça, laquery maintenant labellisée est ajoutée aux autres données labellisées pour être utilisée dans un nouveau cycle d'entraînement.
La boucle d'entraînement-inférence-sélection-labellisation est répétée jusqu'à ce qu'un critère spécifique, comme un coût de labellisation (c-à-d, un nombre maximum de labels donnés par l'oracle) ou une performance minimale du modèle (comme la précision). En faisant cela, on peut attendre une performance décente du modèle tout en labellisant le plus efficacement ses données (merci, apprentissage actif !)
Que fait ALAMBIC ?
ALAMBIC offre une plateforme où tout le processus d'active learning est complètement implémentée avec peu voire même aucun code !
Il suffit juste de:
- Importer ses données ((non-)labellisées)
- Choisir son modèle, ses paramètre et les features à utiliser
- Choisir si on veut entraîner un modèle avec l'apprentissage actif ou étudier le processus de l'apprentissage actif
- Lancer le tout !
Le processus est automatisé avec des visualisation pour suivre la performance du modèle. Vous avez juste besoin de jouer l'oracle !
Comment puis-je l'utiliser ?
Le GitHub repository est en lien en dessous et la documentation complète du projet peut être trouvée sur https://trusted-ai-labs.github.io/ALAMBIC/.
Global explanation landscape of 2D data embeddings using multiple local explanations
(abstract from the paper: https://ceur-ws.org/Vol-3318/short23.pdf)
Nonlinear dimensionality reduction (NLDR) algorithms such as 𝑡-SNE are often employed to visually analyze high-dimensional (HD) data sets in the form of low-dimensional (LD) embeddings. Unfortunately, the nonlinearity of the NLDR process prohibits the interpretation of the resulting embeddings in terms of the HD features. State-of-the-art studies propose post-hoc explanation approaches to locally explain the embeddings. However, such tools are typically slow and do not automatically cover the entire LD embedding, instead providing local explanations around one selected data point at a time. This prevents users from quickly gaining insights about the general explainability landscape of the embedding. This paper presents a globally local and fast explanation framework for NLDR embeddings. This framework is fast because it only requires the computation of sparse linear regression models on subsets of the data, without ever reapplying the NLDR algorithm itself. In addition, the framework is globally local in the sense that the entire LD embedding is automatically covered by multiple local explanations. The different interpretable structures in the embedding are directly characterized, making it possible to quantify the importance of the HD features in various regions of the LD embedding. An example use-case is examined, emphasizing the value of the presented framework.
